Page 73 - 201901
P. 73
第 1 期 仝建波,等: HEPT 类 HIV-1 逆转录酶衍生物的 CoMFA、CoMSIA 及 HQSAR 分析 ·59·
续表 1
No. R 1 R 2 R 3 X pEC 50 No. R 1 R 2 R 3 X pEC 50
27 乙基 硫代苯基 异丙基 氧 6.47 54 甲基 苯基硫代 甲基 硫 5.66
28 甲基 硫代苯基 乙基 氧 6.48 55 甲基 3,5-二氯苯基硫代 2-羟乙基 氧 5.89
3,5-二甲基
29 甲基 2-羟乙基 氧 6.59 56* 乙基 苯基硫代 环己基甲基 硫 6.45
硫代苯基
30 乙基 硫代苯基 异丙基 硫 6.66 57 乙基 苯基硫代 2-羟乙基 硫 6.96
31 乙基 硫代苯基 2-羟乙基 氧 6.92 58 环丙基 苯基硫代 乙基 硫 7.02
32 环丙基 硫代苯基 乙基 氧 7.00 59 乙基 苯基硫代 乙基 氧 7.72
33 乙基 硫代苯基 2-环己基乙基 氧 7.02 60 乙基 3,5-二氯苯基硫代 乙基 硫 7.89
34 甲基 硫代苯基 苄基 氧 7.06 61* 异丙基 苯基硫代 乙基 氧 7.99
35 乙基 硫代苯基 4-甲基苄基 硫 7.00 62 乙基 3,5-二甲基苯基硫代 2-羟乙基 硫 8.11
36 异丙基 硫代苯基 2-羟乙基 氧 7.20 63 乙基 3,5-二甲基苯基硫代 乙基 氧 8.24
37 乙基 3,5-二氯硫代苯基 2-羟乙基 硫 7.37 64 乙基 3,5-二甲基苯基硫代 苄基 氧 8.55
38 乙基 硫代苯基 乙基 硫 7.58 65* 甲基 2-硝基苯基硫代 2-羟乙基 氧 3.85
39 乙基 3,5-二氯硫代苯基 2-羟乙基 氧 7.85 66* 甲基 3-硝基苯基硫代 2-羟乙基 氧 4.47
40 异丙基 硫代苯基 乙基 硫 7.89 67* 甲基 3-碘苯基硫代 2-羟乙基 氧 5.00
41 乙基 硫代苯基 4-氯苄基 硫 7.92 68* 甲基 3-乙酰苯基硫代 2-羟乙基 氧 5.14
42 乙基 硫代苯基 苄基 硫 8.09 69* 甲基 3-溴苯基硫代 2-羟乙基 氧 5.24
43 乙基 3,5-二氯硫代苯基 乙基 氧 8.13 70* 碘代 苯基硫代 2-羟乙基 氧 5.44
44 异丙基 硫代苯基 苄基 硫 8.14 71* 甲基 3-甲基苯基硫代 2-羟乙基 氧 5.59
45 乙基 硫代苯基 苄基 氧 8.23 72 乙烯基 苯基硫代 2-羟乙基 氧 5.69
46 异丙基 3,5-二甲基硫代苯基 2-羟乙基 硫 8.30 73* 甲基 苯基硫代 2-氟乙基 氧 5.96
47 异丙基 硫代苯基 苄基 氧 8.51 74* 甲基 3,5-二甲基苯基硫代 2-羟乙基 硫 6.66
48 异丙基 3,5-二甲基硫代苯基 2-羟乙基 氧 8.57 75* 乙基 苯基硫代 2-苯基乙基 硫 7.04
49 甲基 3-三氟甲基苯基硫代 2-羟乙基 氧 4.35 76* 异丙基 苯基硫代 2-羟乙基 硫 7.23
50 甲基 3-氯苯基硫代 2-羟乙基 氧 4.89 77* 乙基 3,5-二甲基苯基硫代 2-羟乙基 氧 7.89
51 丙基 苯基硫代 2-羟乙基 硫 5.00 78* 乙基 3,5-二甲基苯基硫代 苄基 硫 8.14
52 甲基 苯基硫代 2-羟乙基 氧 5.15 79* 乙基 3,5-二甲基苯基硫代 乙基 硫 8.30
53 甲基 3-氟苯基硫代 2-羟乙基 氧 5.48
注:*为 test 集。
1.2 分子叠合
分子叠合是 QSAR 建模至关重要的一步,叠合
结果对模型的预测精准性以及等势图分析的可靠性
有很大的影响 [15] 。本文选择活性最高、能量最低的
48 号化合物〔6-(3,5-二甲基硫代苯基)-1-(2-
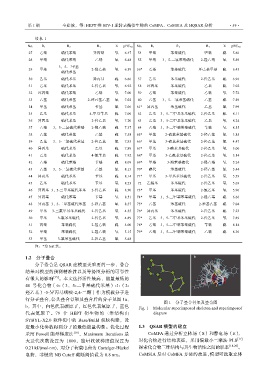
羟乙基)-5-异丙基嘧啶-2,4-二酮〕作为模板分子进
行分子叠合,公共叠合骨架及叠合后的分子见图 1a、
图 1 分子叠合骨架及叠合图
b,其中,白色代表碳原子,红色代表氧原子,蓝色 Fig. 1 Molecular superimposed skeleton and superimposed
代表氮原子。79 个 HEPT 衍生物的三维结构由 diagram
SYBYL-X2.0 软件包中的 Base/Build 模块构建,设
定最小化参数得到分子的最低能量构象。优化过程 1.3 QSAR 模型的建立
采用 Powell 能量梯度法 [16] ,Maximum Iterations 最 CoMFA 通过分析立体场(S)和静电场(E),
大迭代次数设置为 1000,能量收敛梯度值设置为 对化合物进行结构表征,采用偏最小二乘法 PLS [17]
0.21 kJ/(molnm),对分子荷载电荷为 Castelger-Hūckel 探索化合物三维结构与其生物活性之间的联系 [18-20] 。
电荷,非键的 NB Cutoff 截断阈值设为 0.8 nm。 CoMSIA 是对 CoMFA 方法的改进,模型可选取立体